Da SARS-CoV-2 alle sue varianti: struttura e trasmissibilità di un virus che si evolve
di Ylenia Nicolini
Al termine del 2019 un nuovo coronavirus, oggi noto come SARS-CoV-2, è comparso nella città di Wuhan (Cina) diffondendosi poi in tutto il mondo. Ma che cosa sono i coronavirus? Sono un gruppo variegato di virus a RNA appartenenti alla famiglia dei Coronaviridae (ordine dei Nidovirales) in grado di infettare gli esseri umani e molti altri vertebrati causando malattie respiratorie, enteriche, epatiche e neurologiche. Il SARS-CoV-2 è, infatti, il responsabile dell’insorgenza della patologia respiratoria denominata COVID-19. Questo nuovo coronavirus non è ovviamente il primo ad aver colpito gli esseri umani attraverso un salto di specie (i.e. zoonosi). Infatti, due coronavirus altamente patogeni di origine zoonotica (il SARS-CoV e il MERS-CoV) sono stati identificati, rispettivamente nel 2003 e nel 2012, come la causa di gravissime infezioni respiratorie: la sindrome respiratoria acuta grave (SARS) e la sindrome respiratoria del Medio Oriente (MERS). Anche se il SARS-CoV-2 condivide il 79% del proprio genoma con il SARS-CoV e il 50% con il MERS-CoV, è molto più trasmissibile. Per questo, lo scorso 11 marzo 2020, l’Organizzazione Mondiale della Sanità (OMS) ha dichiarato ufficialmente lo stato di pandemia.
Stuttura di SARS-CoV-2
Da quali elementi è composto il SARS-CoV-2?
- RNA a singolo filamento: i virus a RNA a singolo filamento, il cui tasso di mutazione è più elevato rispetto a quello dei virus a DNA, hanno una maggiore capacità di adattamento.
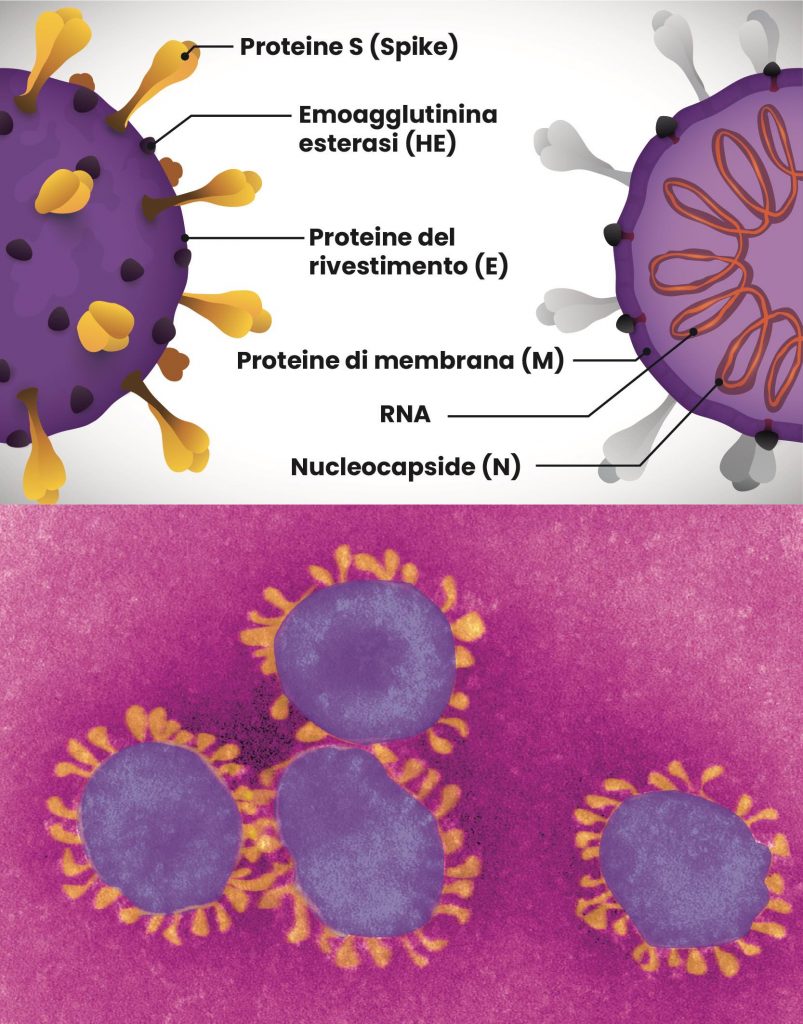
- Un nucleocapside (N): composto dall’RNA genomico e da proteine fosforilate in grado di interagire con la membrana virale.
- Un pericapside (envelope): un doppio strato di fosfolipidi in cui sono immerse diverse proteine strutturali necessarie per l’infezione e la replicazione; tra queste, le proteine di membrana (M) e le proteine del rivestimento (E) sono coinvolte nell’assemblaggio del virus, le proteine S (Spike) e le emoagglutinina esterasi (HE) mediano l’ingresso e la fusione del virus con la cellula bersaglio.
La particella virale, del diametro di circa 60-100 nm, appare rotonda o ovale. Inoltre, se osservata al microscopio elettronico, presenta protuberanze superficiali (formate dalle proteine S) che richiamano la forma di una corona (da cui il nome “coronavirus”) (Figura 1).
Un po’ di epidemiologia, una questione di indici
Quando si cerca di controllare la diffusione di una malattia infettiva emergente è necessario conoscere alcuni parametri epidemiologici chiave, come l’R0. Che cos’è l’R0? L’R0 indica il tasso di riproduzione di una malattia infettiva, ovvero il numero medio di infezioni secondarie trasmesse da ogni individuo infetto all’inizio dell’epidemia, in una fase in cui in genere non sono effettuati interventi per il controllo del fenomeno infettivo. L’R0 rappresenta quindi la trasmissibilità di una malattia infettiva non controllata. L’R0 relativo al SARS-CoV-2 stimato inizialmente dall’OMS nel mese di gennaio 2020 era di circa 1,4-2,5; tuttavia, i dati aggiornati forniti da studi più recenti indicano un R0 maggiore di 4,00. Sulla base di questi risultati, ogni caso di COVID-19 causerebbe almeno altri 3-4 nuovi casi.
L’R0, in sostanza, indica la potenziale trasmissibilità della malattia, ossia il tasso di trasmissibilità “teorico”.
In epidemiologia si utilizza anche l’Rt (indice di trasmissione nazionale) che è nei fatti lo stesso indicatore di trasmissibilità ma calcolato sull’andamento dei contagi anche e soprattutto dopo l’applicazione delle misure di contenimento dell’epidemia stessa: distanziamento sociale, campagne di vaccinazioni (se disponibile un vaccino), dispositivi di protezione individuale, miglioramento delle condizioni igieniche ecc. Il valore dell'Rt infatti si modifica nel tempo seguendo la situazione contingente.
SARS-CoV-2: una nuova variante d’oltremanica
A fine settembre, nel Regno Unito, è stato sequenziato il genoma di un nuovo ceppo di SARS-CoV-2. Si tratta della variante VUI-202012/01 (VUI sta per variant under investigation) che secondo il ministero della Salute inglese avrebbe portato ad un aumento inaspettato del numero di nuovi casi. Un’ipotesi che però necessita di ulteriori conferme epidemiologiche, così come risulta al momento infondato l’allarme per una possibile perdita di efficacia dei nuovi vaccini per COVID-19. Non solo, a detta del consorzio inglese che sta monitorando l’evoluzione del virus, VUI-202012/01 non sembrerebbe causare una malattia più severa.
Dalla prima sequenza genomica di SARS-CoV-2 conosciuta e depositata a inizio gennaio a oggi sono moltissime le mutazioni che si sono generate. Quando le mutazioni si accumulano nel tempo il virus può cambiare parzialmente le proprie caratteristiche al punto tale da dare origine a una variante virale rispetto al virus originale. Ed è questo il caso di VUI-202012/01 che a Londra e nel Kent rappresentava il 28% dei casi a metà novembre e il 62% la settimana del 7 dicembre, diventando il ceppo prevalente in poche settimane. VUI-202012/01 presenta diverse mutazioni, ma tre sono gli aspetti che hanno attirato l’attenzione degli esperti: si sta rapidamente sostituendo ad altre varianti del virus; ha mutazioni che interessano la proteina Spike (coinvolta nel processo di fusione con le cellule umane); alcune di queste mutazioni sono già state isolate in laboratorio e dimostrano capacità maggiori di infettare altre cellule. Tutti questi elementi concorrerebbero ad aumentare la capacità di trasmissione del virus. L’OMS ha, infatti, affermato l’esistenza di “segnali preliminari che indicano come il virus sia in grado di diffondersi più facilmente tra le persone e possa influenzare le prestazioni di alcuni test diagnostici”. Gran parte della comunità scientifica considera però improbabile che i vaccini, in procinto di essere distribuiti in tutta Europa, risultino inefficaci contro la nuova variante. Infatti, grazie al vaccino il sistema immunitario produce moltissimi anticorpi di tipo differente in grado di riconoscere diverse porzioni della proteina Spike. Qualora una mutazione modifichi una specifica porzione di proteina tale da non essere riconosciuta dagli anticorpi, esistono altri anticorpi capaci di riconoscere tutte le restanti parti della proteina. Per questa ragione la possibilità che i vaccini appena sviluppati non siano efficaci è remota e comunque tutta da dimostrare.
Sitografia:
COVID-19 Animation: What Happens If You Get Coronavirus?
Covid-19 Genomics UK Consortium
No evidence for increased transmissibility from recurrent mutations in SARS-CoV-2, Nature
Bibliografia:
Boopathi, S., Poma, A. B., & Kolandaivel, P. (2020). Novel 2019 coronavirus structure, mechanism of action, antiviral drug promises and rule out against its treatment. Journal of Biomolecular Structure and Dynamics, 1-10.
D’Arienzo, M., & Coniglio, A. (2020). Assessment of the SARS-CoV-2 basic reproduction number, R0, based on the early phase of COVID-19 outbreak in Italy. Biosafety and Health.
Hu, B., Guo, H., Zhou, P., & Shi, Z. L. (2020). Characteristics of SARS-CoV-2 and COVID-19. Nature Reviews Microbiology, 1-14.
Jiang, F., Deng, L., Zhang, L., Cai, Y., Cheung, C. W., & Xia, Z. (2020). Review of the clinical characteristics of coronavirus disease 2019 (COVID-19). Journal of general internal medicine, 1-5.
Ke, Z., Oton, J., Qu, K., Cortese, M., Zila, V., McKeane, L., ... & Lu, J. M. (2020). Structures and distributions of SARS-CoV-2 spike proteins on intact virions. Nature, 1-7.
Li, F. (2016). Structure, function, and evolution of coronavirus spike proteins. Annual review of virology, 3, 237-261.
Mousavizadeh, L., & Ghasemi, S. (2020). Genotype and phenotype of COVID-19: Their roles in pathogenesis. Journal of Microbiology, Immunology and Infection.
Zhang, S., Diao, M., Yu, W., Pei, L., Lin, Z., & Chen, D. (2020). Estimation of the reproductive number of novel coronavirus (COVID-19) and the probable outbreak size on the Diamond Princess cruise ship: A data-driven analysis. International journal of infectious diseases, 93, 201-204.















